今天我们就来介绍用来查询融合基因功能的数据。
1 数据库分析流程
由于要分析融合基因的功能,所以第一步就是要选择分析哪些融合基因的功能。这里作者纳入了三个数据库当中和肿瘤相关的融合基因。这三个数据库分别是ChiTaRS 3.1、TumorFusions以及一篇基于TCGA分析融合基因文章(pmid: 29617662)。最终收集到了48000个融合基因,进一步作者对这这些融合基因进行肿瘤相关的功能注释。

2 主要注释内容
对于这些的融合基因,作者进行了多种功能的注释。
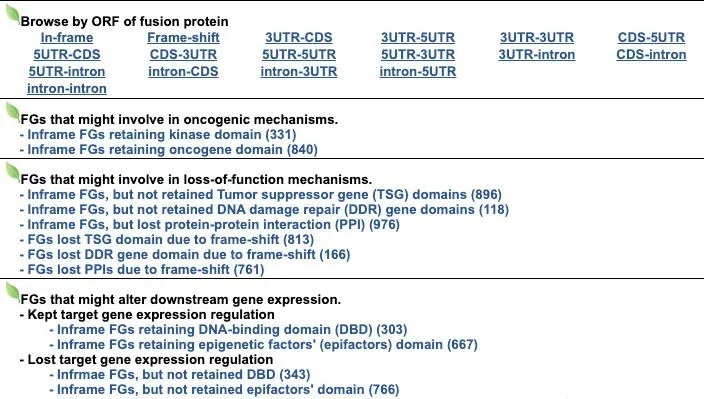
通过开放阅读框进行功能注释。融合基因通过开放阅读框注释成不同的类型。
对可能致癌的机制进行了注释。
对其基因功能变化进行了注释。
以及对下游的基因表达的变化的注释。
3 数据库使用
对于数据库的使用而言,比较简单,我们输入我们想要查看的和某一个基因有关的融合基因即可。例如我们这里输入: ABL1.
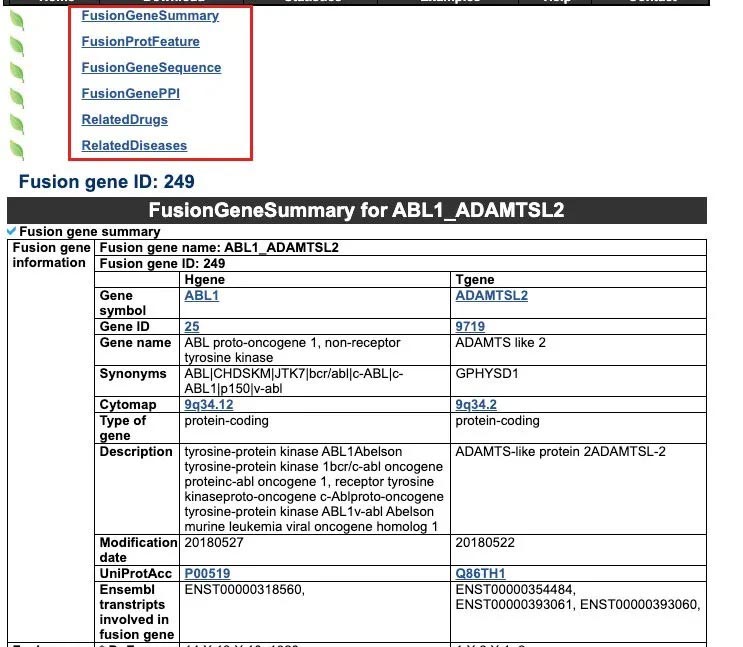
首先,我们能看到的是和这个基因有关的融合基因。我们点击我们想要查看的融合基因的ID号就可以查看其具体的信息。例如我们选择: ABL1_BCR。
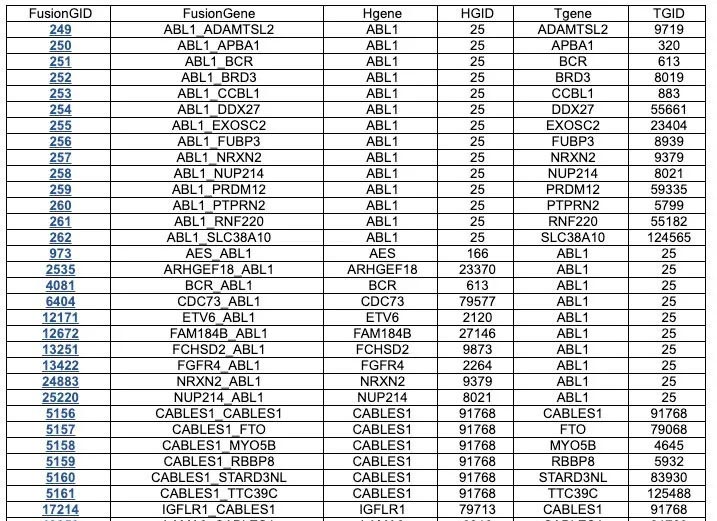
结果的展示出了相关基因的具体信息,我们还可以查看融合基因蛋白相互作用、药物相关信息以及和基因相关的信息。
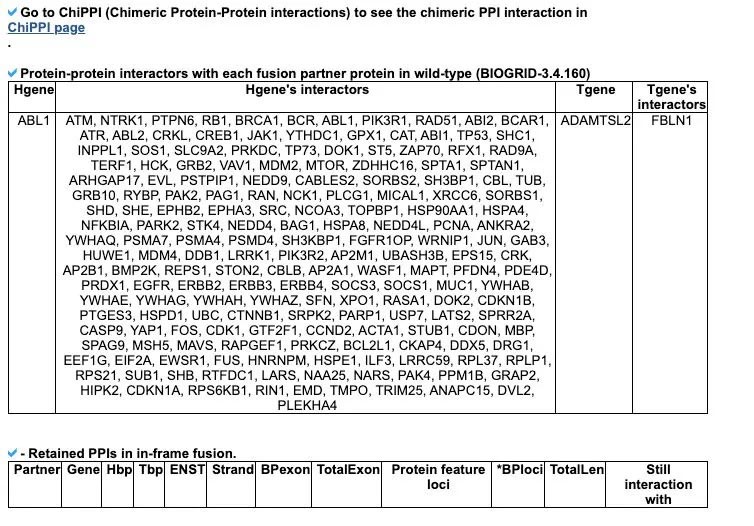
其中关于融合基因蛋白相互作用的预测是基于BIOGRID(v 3.4.260)数据的结果来的。
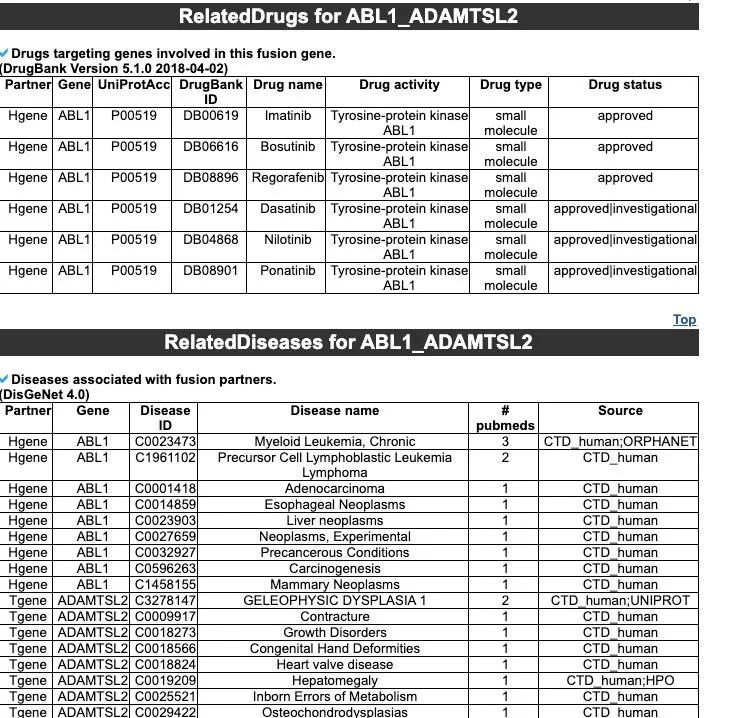
关于融合基因药物和疾病相关的数据来自于DrugBank和DisGeNet。
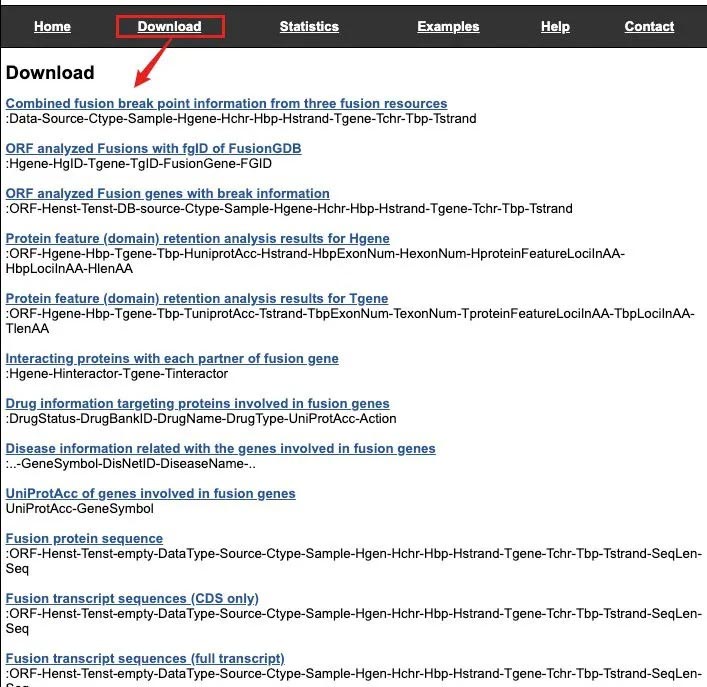
4 数据下载
另外这个数据库提供了所有相关数据的下载,如果想要查看某一个方面的所有数据库的话,可以来下载具体的数据。